(篇幅较长,耐心读,一定受益匪浅)
研究目的与意义:1974年首次发现m6A甲基化,它是一种RNA分子上的甲基化修饰,m6A修饰在哺乳动物细胞中是动态可逆的,类似于DNA和组蛋白修饰的另一种表观遗传调控,近年来m6A甲基化已成为生命科学领域的研究热点。通过诸多学者辛苦研究,结果表明在mRNA中发现的m6A修饰可以调节癌细胞的生命活动。另外我们知道EMT是上皮细胞获得间充质细胞特质的一种重要现象,通过EMT诱导转录因子(例如Snail,Slug,Twist和Zeb)来抑制e-cadheri的表达促进癌细胞的转移,此外,Snail是e-cadherin表达的重要转录因子,近年来围绕EMT的表观遗传调控因素做了诸多研究,发现DNA甲基化及组蛋白修饰等均可参与肿瘤细胞EMT的发生发展,但mRNA修饰对肿瘤细胞EMT的影响尚未揭示。
一、癌细胞中EMT受mRNA的甲基化水平调控
以上所有这些数据表明用TGF-β处理的癌细胞正在进行EMT过程。
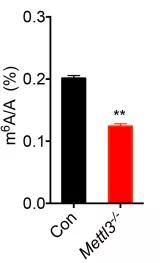
为了证明m6A在EMT过程中的作用,作者使用CRISPR / Cas9编辑系统,在HeLa细胞中敲低METTL3,形成 METTL3Mut/− HeLa细胞。下图是METTL3在野生型及METTL3Mut/− HeLa细胞中的蛋白表达情况。
进一步通过LC-MS/MS分析,结果显示,METTL3 Mut/− HeLa 细胞的m6A水平显着低于野生型细胞,这也证实了METTL3作为mRNA的m6A具有介导催化作用。

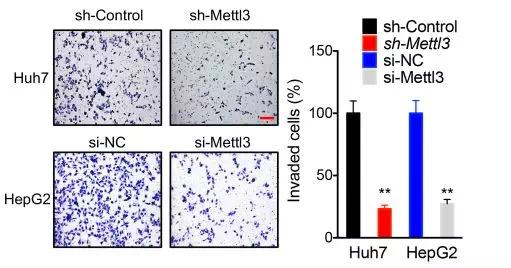
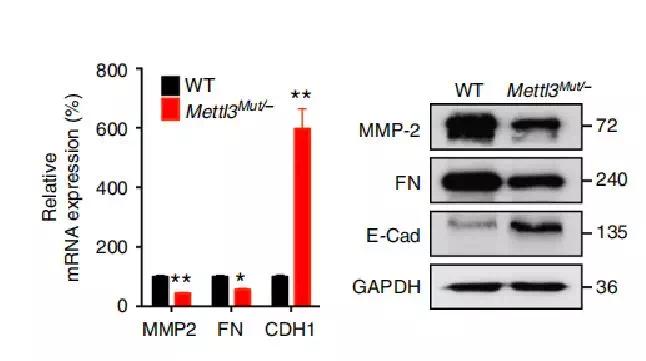
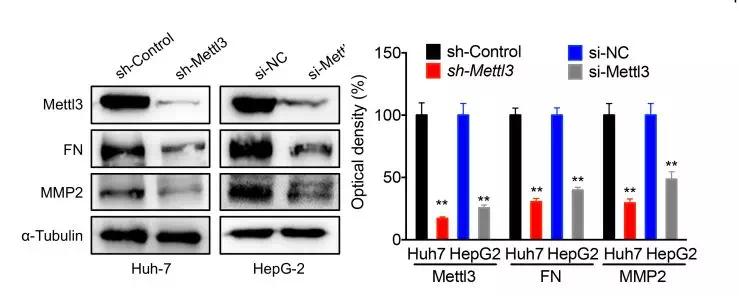
这些数据表明METTL3的缺失可以抑制癌细胞EMT的发生;
进一步通过蛋白质印迹分析显示ALKBH5的过表达增加了E-Cad,同时降低了HeLa细胞中的MMP2和FN。
此外,在METTL3Mut/−HeLa和Mett13敲低的Huh7细胞中恢复了由TGF-β诱导的E-Cad下调和FN上调。
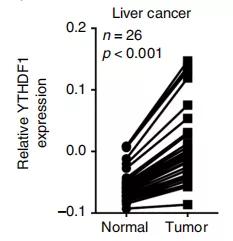
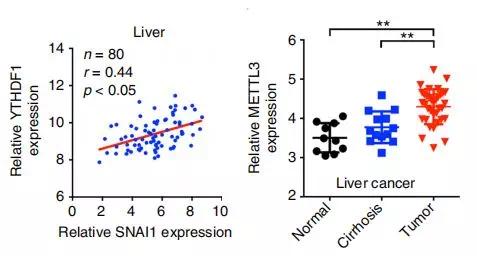
根据上述数据已经知道METTL3的缺失可以抑制癌细胞EMT的发生,作者进一步研究了METTL3的临床作用。TCGA数据显示,METTL3在肝癌组织中的表达显着高于癌旁组织。
在364名肝癌患者中,METTL3的表达与CDH1 mRNA呈负相关。这种关联与上述数据呈现结果一致 。
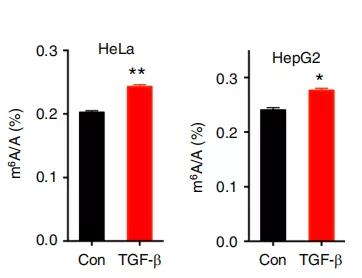
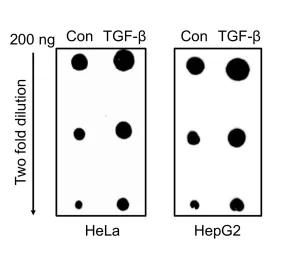
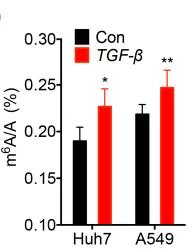
根据Bertero等的报道,TGF-β可通过Smad2 / 3与METTL3-METTL14- WTAP复合物之间的相互作用诱导人胚胎干细胞(hESCs)中的mRNA甲基化。通过使用Smad2 / 3抑制剂B431542(10μM)预处理HeLa细胞,然后用TGF-β进一步处理3天,通过LC-MS/MS测定总mRNA的m6A 与A的比例,来计算m6A在mRNA上整体的甲基化程度。发现Smad2/3抑制剂SB431542(SB)可以阻碍TGF-β诱导的HeLa细胞中m6A的上调。

二、EMT的细胞,m6A会调控其它基因的变化
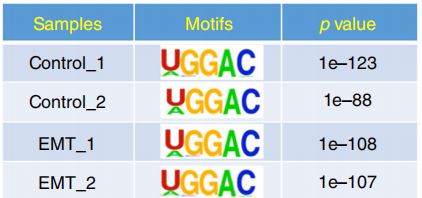

总的来说,m6A-seq数据显示m6A修饰发生在EMT期间与细胞连接,粘附和迁移相关的少数基因上。
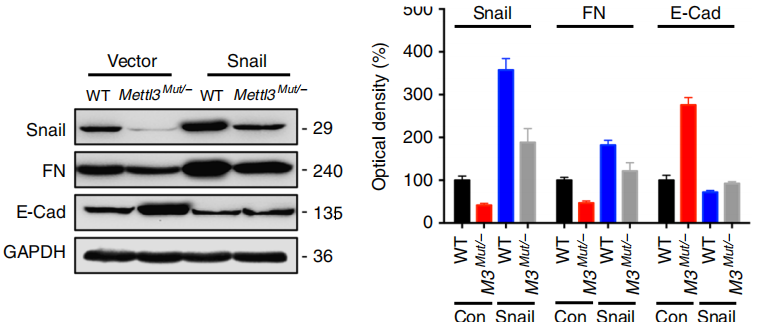
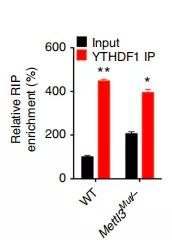
为了验证Sainl是参与m6A调节的癌细胞EMT的潜在靶点,作者通过在EMT期间鉴定84个EMT的相关基因和61 个m6A调节基因(大于2倍m6A变化),最终确定了四个重叠基因。分别为SANI1、CTGF、MSN、CTNNB1,在四种鉴定的基因中,Snail被认为是控制EMT的重要转录因子。作者的数据证实Sainl mRNA被m6A修饰,并且在EMT进展期间在CDS和3'UTR区域中m6A的显着富集,另外,作者通过RIP技术,发现在EMT细胞中,m6A水平的Sainl-mRNA显著增加,富集倍数与对照相比约为2.3倍。
接下来,作者研究了在HeLa细胞中敲低METTL3、以及过表达ALKBH5时Snail蛋白的表达情况,。在METTL3缺失和ALKBH5过表达的两种情况下,作者观察到HeLa和HepG2细胞中Snail蛋白水平的降低而ZEB1表达没有变化。
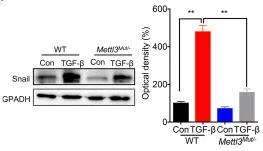
为了证实m6A对Snail表达有作用,作者用TGF-β处理野生型和METTL3Mut /-HeLa细胞。结果显示,在METTL3Mut /-HeLa细胞中,TGF-β诱导的Snail表达低于对照细胞,这表明m6A参与TGF-β诱导的癌细胞中Snail的表达。
四、m6A促进癌细胞中Snail mRNA的翻译
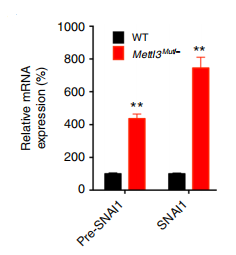
用Act-D处理野生型和METTL3Mut /-HeLa细胞以阻断转录。结果显示野生型细胞中pre-mRNA和mat-mRNA的半衰期显着短于METTL3Mut /-HeLa 细胞。表明m6A修饰可能引发pre-mRNA的剪切和癌细胞中Snail mat-mRNA的降解。
另外作者补充证实,Snail是YTHDF2的靶标,其负责m6A介导的转录物的mRNA去稳定化。过量表达YTHDF2可降低HeLa细胞中成熟Snail mRNA的表达和稳定性;
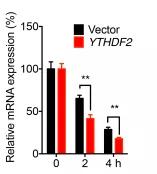

此外,作者用MG132预处理METTL3Mut /-HeLa细胞以抑制蛋白体外酶活性和使用CHX阻断蛋白翻译,然后用TGF-β处理。结果数据显示,在CHX而非MG132存在下, TGF-β诱导的METTL3Mut /-HeLa细胞中的Snail表达减弱。
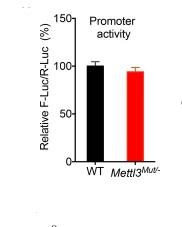
随后作者将Snail CDS连接到多克隆位点(MCS)构建了pmirGLO-Snail荧光素酶报告基因。 双荧光素酶测定显示,Snail在METTL3Mut /-HeLa细胞中的翻译效率显着低于野生型细胞。
下面作者对RNA进行分类,通过多聚核糖体分析,发现METTL3Mut /-HeLa细胞中80S的核糖单体和多聚核糖体都低于野生型细胞。
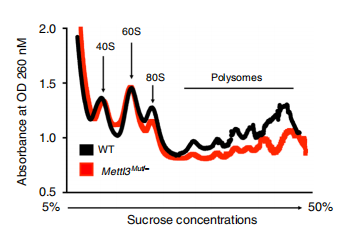
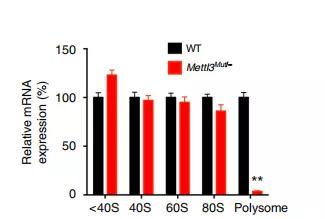

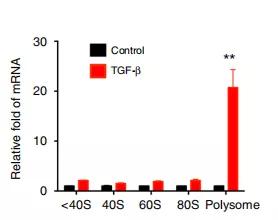 表明在EMT过程促进SNAI1 mRNA的转录翻译。
表明在EMT过程促进SNAI1 mRNA的转录翻译。
作者通过对m6A-RIP测序,数据显示CDS区域中m6A峰值位于第20条染色体上,48,600,490 to 48,600,630且在3 'UTR。
作者鉴定了3个GGAC motif,分别位于CDS区域和Snail mRNA的3'UTR区域;
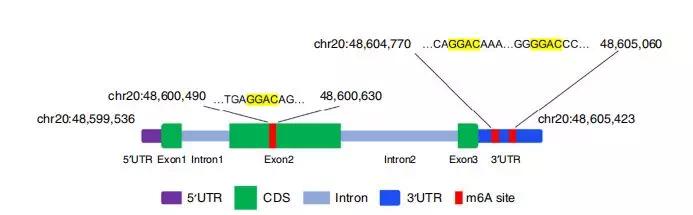
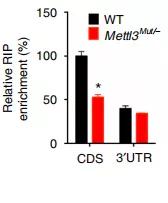
下面作者对m6A甲基化对SNAI13'UTR及CDS区域分别作了详细研究。
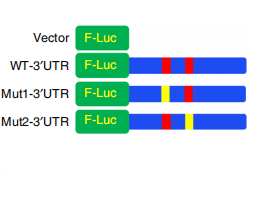
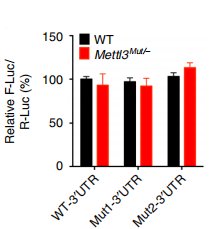
这表明3'UTR上的m6A甲基化可能不参与m6A修饰调节的Snail表达;

数据显示,在过量表达pcDNA-Snail-CDS-mut1的METTL3Mut细胞中部分上调了Snail表达水平(上图)。将pcDNA-Snail-CDS-WT、pcDNA-Snail CDS-mut1 / mut2转染的HeLa细胞进行TGF-β处理。 蛋白印迹结果显示,TGF-β诱导的pcDNA Snail-CDS-mut1的Snail表达低于pcDNA-Snail-CDS-WT和pcDNA-Snail-CDS-mut2(下图)。
总之,结果数据表明SNAI1 CDS区域中的m6A是表达调控的主要位置;
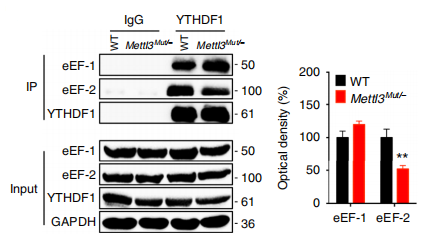
由于真核生物的翻译延伸是由两个延伸因子作用进行的,:eEF-1和eEF-2,作者观察了MetE13Mut -HeLa细胞中eEF-1和eEF-2结合Snail mRNA的变异。数据显示,在METTL3Mut-HeLa细胞中Snail mRNA和eEF-2之间的结合显着低于HeLa细胞中的结合。 此外,通过蛋白定性分析表明,得到了类似的结果。
六、m6A与癌细胞发展有关

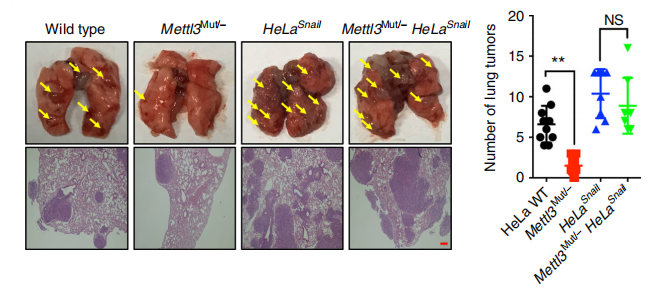
这表明Snail参与了METTL3的体内转移作用。













 首页
首页
 关于我们
关于我们
 产品中心
产品中心
 仪器中心
仪器中心
 新闻中心
新闻中心
 资料下载
资料下载
 在线留言
在线留言
 人才招聘
人才招聘
 联系我们
联系我们